无监督学习顾名思义数据中不包含已知的输出结果,学习算法中只有输入数据,算法需要从这些输入数据中提取相关规律。 无监督学习主要分为两种类型:数据集变换与聚类算法,数据集的无监督变换是创建数据集的新的表达方式,使其特性更容易理解,最常见的模型有 PCA、NMF、t-SNE 等模型。聚类算法则是将数据划分成不同的组,每组数据中包含有类似的特征,常见的模型有 KMeans、DBSCAN、GMM、Agglomerative 等,下面将对各种模型的特性与应用场景作详细介绍。
数据集变换的一个主要常见应用就是降维和压缩数据,从多维数据中提取其重大的特征,最常用的模型就是 PCA 与 NMF。 另一个应用是流形学习,它尝试把一个低维度流形数据嵌入到一个高维度空间来描述数据集,通过转换找不到的数据规律,常见的模型有 t-SNE、MDS、LLE、Isomap 等。
一、PCA 主成分分析
1.1 PCA 降维的基本原理
主成分分析 PCA(Principal Component Analysis)是最常用的非监督学习,常用于高维数据的降维,可用于提取数据的主要特征分量。PCA 常常与监督学习合并使用,特别是在数据量较大的情况下,通过 PCA 压缩数据集,再使用监督学习模型进行分析可提升系统有效性。 PCA 会根据设置把数据点分解成一些分量的加权求和,利用降维的思想,把多指标转化为少数几个综合指标。这个变换把数据变换到一个新的坐标系统中,使得任何数据投影的第一大方差在第一个坐标(称为第一主成分)上,第二大方差在第二个坐标(第二主成分)上,依次类推。主成分分析常常用于减少数据集的维数,同时保持数据集的对方差贡献最大的特征。
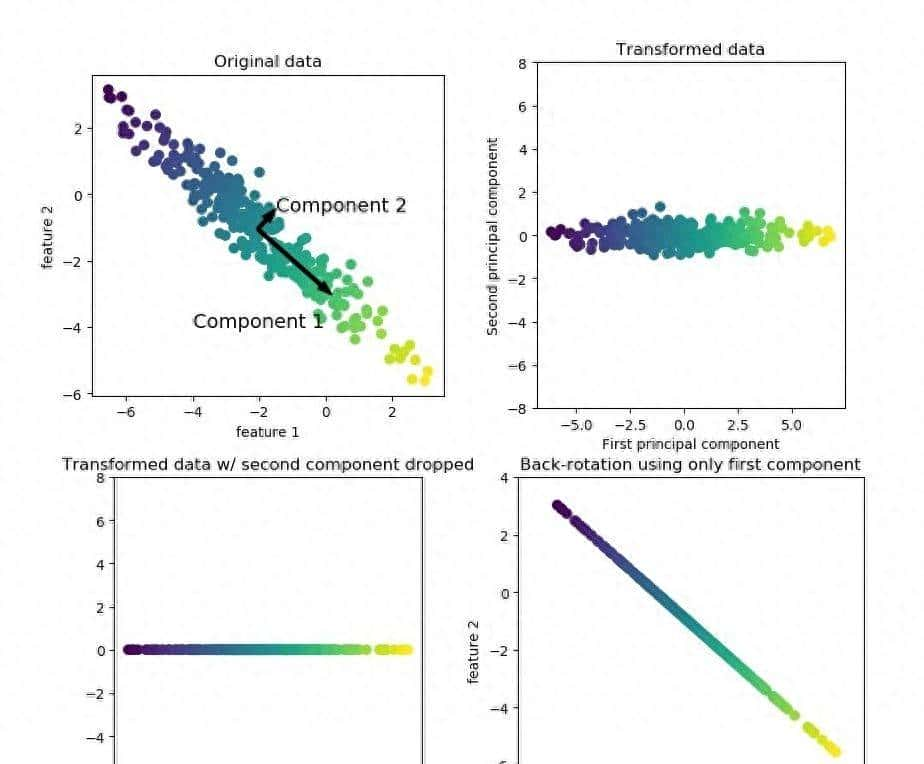
下面以一个最简单的例子作为说明,如图以一个二维特征的数据集为例,PCA 使用旋转数据集的方法,把旋转后新数据特征的重大性来选择特征的子集。左上角的图是原始的数据点,算法先找到方差最大的方向 Component1,此方向包含最多的信息量。然后并以其垂直方向为 Component 2,如此类推,由于这是二维数据集所以只包含两个成分,一般主成分个数会与维度数相等。定好成分后来,如图二把坐标轴旋转,令第一个成分与 X 轴平行,第二个成分与 Y 轴平行。在旋转时从数据中减去平均值,使到转换后的数据都是以 0 为中心。由于 X 轴与 Y 轴是不相关的,所以除了对角线,其他的矩阵都为 0。因此在图三旋转后只保存了 Component1 成分,这就把数据集从二维数据集降到一维数据集。最后把轴重新旋转到原来方向,并把平均值加到数据中,就得到图四。这就是最简单的 PCA 降维过程。
1.2 PCA 类简介
构造函数
1 class PCA(_BasePCA):
2 @_deprecate_positional_args
3 def __init__(self, n_components=None, *, copy=True, whiten=False,
4 svd_solver='auto', tol=0.0, iterated_power='auto',
5 random_state=None):
6 ......参数说明
- n_components:int, float 类型 or 'mle', 默认值国 None。int 时则是直接指定希望PCA降维后的特征维度数目,此时 n_components是一个大于等于1的整数。也可以使用 float 指定主成分的方差和所占的最小比例阈值,让PCA类自己去根据样本特征方差来决定降维到的维度数,此时 n_components是一个[0,1]之间的数。当然,我们还可以将参数设置为”mle”, 此时PCA类会用MLE算法根据特征的方差分布情况自己去选择必定数量的主成分特征来降维。我们也可以用默认值,即不输入n_components,此时n_components=min(样本数,特征数)。
- copy: bool 类型,表明是否在运行算法时,将原始数据复制一份。默认为True,则运行PCA算法后,原始数据的值不会有任何改变。由于是在原始数据的副本上进行运算的。
- whiten :bool 类型,判断是否进行白化,默认值为 False ,即不进行白化。所谓白化,就是对降维后的数据的每个特征进行归一化,让方差都为1.对于PCA降维本身来说,一般不需要白化。如果你PCA降维后有后续的数据处理动作。
- svd_solver:str 类型,可选值 {'auto', 'full', 'arpack', 'randomized'} 之一,默认值为 auto 。即指定奇异值分解SVD的方法,由于特征分解是奇异值分解SVD的一个特例,一般的PCA库都是基于SVD实现的。randomized一般适用于数据量大,数据维度多同时主成分数目比例又较低的PCA降维,它使用了一些加快SVD的随机算法。 full则是传统意义上的SVD,使用了scipy库对应的实现。arpack和randomized的适用场景类似,区别是randomized使用的是scikit-learn自己的SVD实现,而arpack直接使用了scipy库的sparse SVD实现。默认是auto,即PCA类会自己去在前面讲到的三种算法里面去权衡,选择一个合适的SVD算法来降维。一般来说,使用默认值就够了。
- tol: float 类型,默认值为 0.0,代表求解方法精度。
- iterated_power: int 类型,默认auto,代表当 svd_solver == ‘randomized’ 时幂方法的迭代次数。
- random_state: int 类型,默认None,随机数种子,推荐设置一个任意整数,同一个随机值,模型可以复现。
常用属性
- components_:返回主要成分,运行 transform 后的主要成分将保存在此属性当中。
- n_components_:返回所保留的成分个数 n。
- explained_variance:特征方差值,方差值越大,说明重大性要越高。与 explained_variance_ratio 同时使用可更清晰分辨每个特征的占比。
- explained_variance_ratio_:每个特征方差贡献率,个比例越大,说明重大性要越高,所有总和为1。
- noise_variance_: 根据概率主成分分析模型估计的噪声协方差。
常用方法
- fit(self, X, y=None): 表明用数据X来训练PCA模型。
- transform(selft,X):将数据X转换成降维后的数据。通过与 fit 同用,先调用 fix,当模型训练好后,再使用 transform 方法来降维。
- fit_transform(self, X, y=None): 用X来训练PCA模型,同时返回降维后的数据。相当于结合了 fit 与 transform 两个方法。
- inverse_transform(self, X):将降维后的数据转换成为原始数据。
1.3 应用实例
1.3.1 使用 PCA 降噪
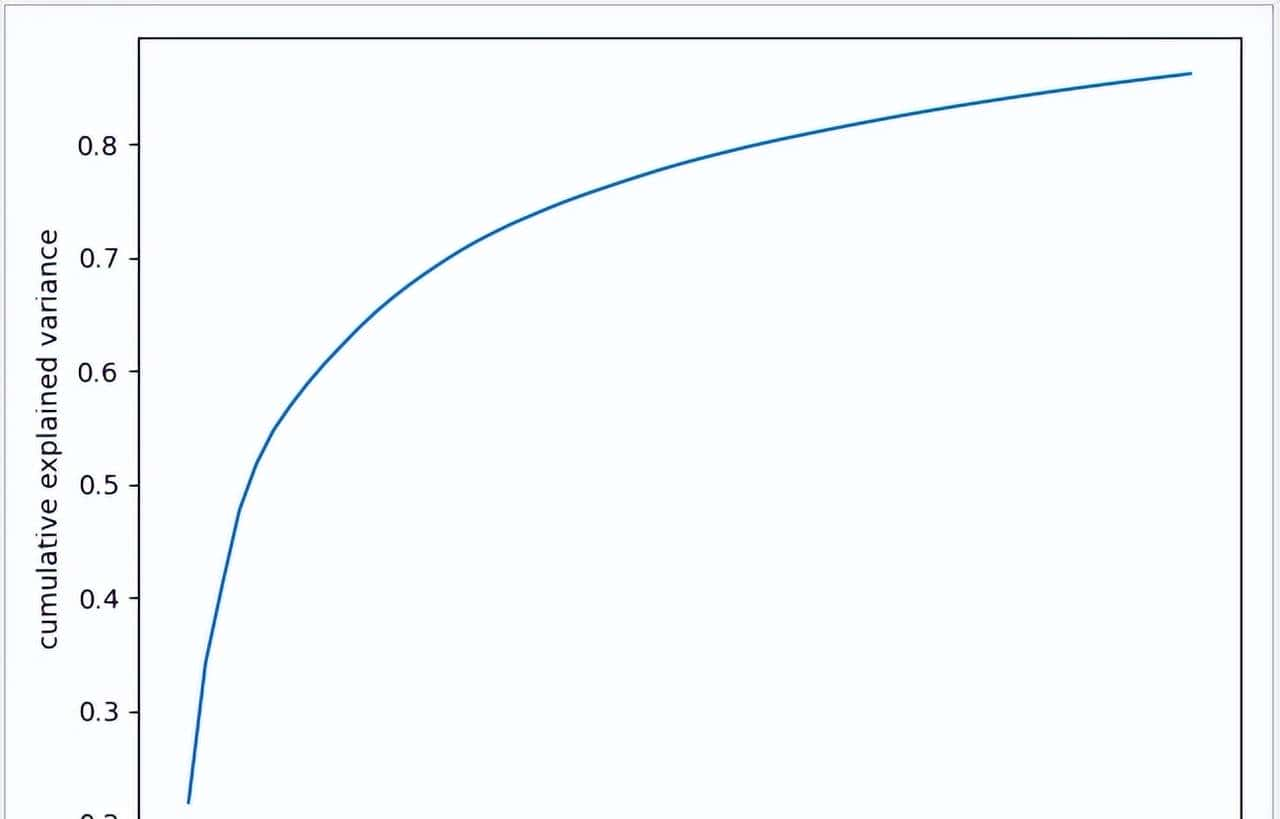
尝试先使用 PCA 降噪,提取 60 个主要成分,再使用 KNeighborsClassifier 模型进行训练,对比一下测试结果。使用 PCA 降噪后,准确率从 30% 上升到 40%,可见通过有效提取主要成分有可能得到更好的数据模式。一般可以通过 explained_variance_ratio_ 属性,来判断所需要成分的数量,成分数量过少不能反映数量集的主要特征,成分数量过多,丢失了 PCA 降燥的意义,一般会把 explained_variance 保持到 90% 以上。
1 def knn_test():
2 #测试数据集
3 person=datasets.fetch_lfw_people(min_faces_per_person=20)
4 X_train,X_test,y_train,y_test=train_test_split(person.data,person.target,random_state=1)
5 #使用 KNeighborsClassifier 模型进行测试
6 knn = KNeighborsClassifier()
7 knn.fit(X_train, y_train)
8 #输出knn准确率
9 print('KNN:
train data:{0}
test data:{1}'
10 .format(knn.score(X_train, y_train),
11 knn.score(X_test, y_test)))
12
13 def pca_test():
14 #测试数据集
15 person=datasets.fetch_lfw_people(min_faces_per_person=20)
16 X_train,X_test,y_train,y_test=train_test_split(person.data,person.target,random_state=1)
17 #建立PCA模型,使用60个主要成分
18 pca=PCA(60 ,whiten=True,random_state=2).fit(X_train)
19 #将数据X转换成降维后的数据
20 X_train_pca=pca.transform(X_train)
21 X_test_pca=pca.transform(X_test)
22 #建立 KNeighborsClassifier 模型
23 knn=KNeighborsClassifier()
24 #把过滤后60个特征的数据放入KNN模型进行训练
25 knn.fit(X_train_pca,y_train)
26 #输出准确率
27 print('
PCA->KNN:
train data:{0}
test data:{1}'
28 .format(knn.score(X_train_pca, y_train),
29 knn.score(X_test_pca, y_test)))
30 #观察累计方差贡献率
31 plt.plot(np.cumsum(pca.explained_variance_ratio_))
32 plt.xlabel('number of components')
33 plt.ylabel('cumulative explained variance')
34 plt.show()
35
36 if __name__ == '__main__':
37 knn_test()
38 pca_test()运行结果

1.3.2 提取面部特征
PCA 通过 fit_transform 方法完成降维后,可通过 inverse_transform 方法将降维后的数据转换成为原始数据。对比一下图一与图二,可以辨别二种不同的情况,通过使用此方法对图片进行压缩,原来 3023 个特征已经被压缩到 175 个,图片的特征已比较清晰。特征成分保存在 PCA 的 components_ 属性中,图三显示了前 15 个特征成分。
1 def pca_test():
2 #测试数据集
3 person=datasets.fetch_lfw_people(min_faces_per_person=20)
4
5 #建立PCA模型,使用 95% 的主要成分
6 pca=PCA(0.95 ,whiten=True,random_state=2)
7 #将数据X转换成降维后的数据
8 componmets=pca.fit_transform(person.data)
9 #显示原图
10 index1,index2,index3=0,0,0
11 fix, axes1 = plt.subplots(3, 5, figsize=(60, 40))
12 for ax in axes1.ravel():
13 a=person.data[index1,:]
14 ax.imshow(person.data[index1,:].reshape(62,47),cmap='viridis')
15 ax.set_title('original '+str(index1),fontsize=100)
16 index1+=1
17 #提取特征成分后还原数据
18 fix, axes2 = plt.subplots(3, 5, figsize=(60, 40))
19 for ax in axes2.ravel():
20 image = pca.inverse_transform(componmets[index2])
21 ax.imshow(image.reshape(62, 47), cmap='viridis')
22 ax.set_title('inverse '+str(index2),fontsize=100)
23 index2 += 1
24 #显示特征成分
25 fix, axes3 = plt.subplots(3, 5, figsize=(60, 40))
26 for ax in axes3.ravel():
27 ax.imshow(pca.components_[index3].reshape(62, 47), cmap='viridis')
28 ax.set_title('component ' + str(index3), fontsize=100)
29 index3 += 1
30 plt.show()运行结果

原图

提取成分后还原图

特征成分图

二、NMF 非负矩阵分解
2.1 NMF 基本原理
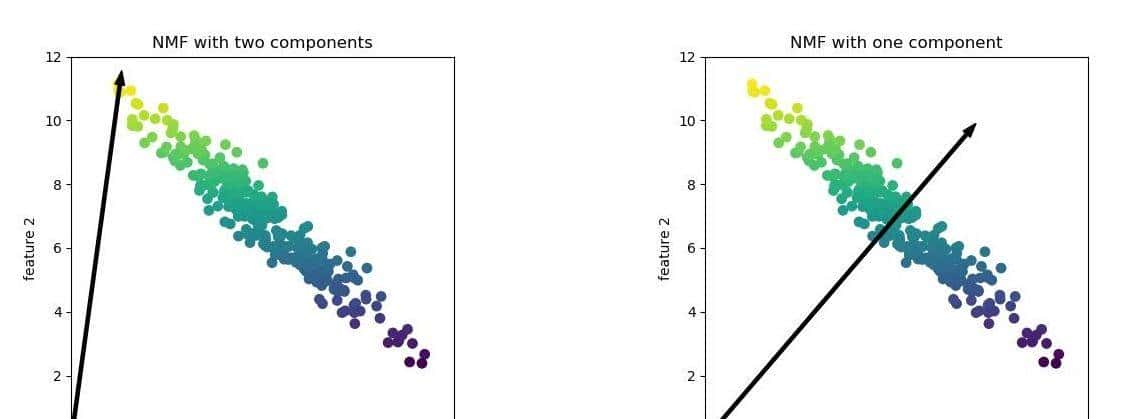
非负矩阵分解 NMF(non-negatie matrix factorization)是另一种用于数据集变换的无监督学习,与 PCA 类似其目在于降维以及提取有用特征。NMF 尝试将每个数据点分解成一些分量的加权求和,不过与 PCA 不同的是,PCA 使用的是正交分量,把数据点解释成为数据方差。而 NMF 中将要分解的系数均为非负值,也就是说所有被分解的系数均大于等于0,因此被分解后的每个特征均为非负值。 NMF 使用了随机初始化,不同的随机种子可能产生不同的结果。对于两个分量的 NMF 如左图,所有的数据点都可以分解成两个正数组合。特征数越多,分量个数越多,只要有足够多的分量,特征就可以完成地分解。如果仅使用一个分量如右图,NMF 就会创建一个指向平均值的分量。
2.2 NMF 类简介
构造函数
1 class NMF(TransformerMixin, BaseEstimator):
2 @_deprecate_positional_args
3 def __init__(self, n_components=None, *, init='warn', solver='cd',
4 beta_loss='frobenius', tol=1e-4, max_iter=200,
5 random_state=None, alpha=0., l1_ratio=0., verbose=0,
6 shuffle=False, regularization='both'):参数说明
- n_components:int, 默认值为 None。int 时则是直接指定 NMF 降维后的特征维度数目,此时 n_components是一个大于等于 1 的整数。当使用默认值,即不输入n_components,此时n_components=min(样本数,特征数)。
- init: str 类型,可选择 {'random', 'nndsvd', 'nndsvda', 'nndsvdar', 'custom'} 之一,默认为 None。选择W,H迭代初值的算法, 当为 None 时,即自动选择值,不使用选择初值的算法。'random':非负随机矩阵,缩放为:sqrt(X.mean() / n_components); ‘nndsvd’`:非负双奇异值分解(NNDSVD),初始化(对于稀疏性更好); 'nndsvda':NNDSVD与零填充了X的平均值(当不需要稀疏性时,效果更好); ' nndsvdar ': NNDSVD带充满小随机值的零(一般替代NNDSVDa 由于当不需要稀疏性时); 'custom':使用自定义矩阵W和H。
- solver: str 类型,默认为 ‘cd' 坐标轴下降法 , 可选 {’cd','mu'} 。 cd 是坐标轴下降法cd,mu 是乘性更新算法。
- beta_loss:float 类型 或 {'frobenius', 'kullback-leibler', 'itakura-saito'} 之一,默认是 ’frobenius’。 衡量V与W H之间的损失值。
- tol: float 类型,默认值为 1e-4,代表求解方法精度。
- max_iter:默认值为 200 ,指定了模型优化的最大迭代次数。
- random_state:int 类型,默认None,随机数种子,推荐设置一个任意整数,同一个随机值,模型可以复现。
- alpha:float 类型,是正则项系数,初始值为 0.0,数值越大,则对复杂模型的惩罚力度越大。
- l1_ratio : float类型, 默认值为 0.0 弹性净混合参数。当 l1_ratio=0 对应于L2惩罚,当 l1_ratio=1对应 L1 惩罚,当0<l1_ratio<1,惩罚是L1和L2的组合。
- verbose: int类型,默认值为0,代表详细程度
- shuffle: bool 类型,默认值为 False, 如果为true,则随机化 CD 求解器中的坐标顺序。
- regularization: str 类型,{'both', 'components', 'transformation', None} 之一 , 默认值 为 'both'。选择正则化是否影响 components(H),transformation(W),both 则两者都有,None 则两者都没有。
2.3 应用实例
2.3.1 NMF 提取特征
用回 PCA 的一样例子进行测试,用 150 个主要成分建立 NMF 模型,提取特征成分后还原数据,把还原后数据和特征成分显示出来。与 PCA 进行对比,可见 NMF 模型的特征与 PCA 提取的特征有明显差别,NMF 更具有人脸特征的原型。
1 def nmf_test():
2 person=datasets.fetch_lfw_people(min_faces_per_person=20)
3 #建立NMF模型,使用 150 个主要成分
4 nmf=NMF(150,random_state=2)
5 #将数据X转换成降维后的数据
6 componmets=nmf.fit_transform(person.data)
7 index1,index2=0,0
8 #提取特征成分后还原数据
9 fix, axes1 = plt.subplots(3, 5, figsize=(60, 40))
10 for ax in axes1.ravel():
11 image = nmf.inverse_transform(componmets[index1])
12 ax.imshow(image.reshape(62, 47), cmap='viridis')
13 ax.set_title('inverse '+str(index1),fontsize=100)
14 index1 += 1
15 #显示特征成分
16 fix, axes2 = plt.subplots(3, 5, figsize=(60, 40))
17 for ax in axes2.ravel():
18 ax.imshow(nmf.components_[index2].reshape(62, 47), cmap='viridis')
19 ax.set_title('component ' + str(index2), fontsize=100)
20 index2 += 1
21 plt.show()运行结果
提取成分后还原图

特征成分图

2.3.2 还原面部特征
从上面例子看到的特征成分图可见 NMF 更具有人脸特征的原型。从 component 6 可以看出人脸是向右侧,而 component 10 是向左侧。目前尝试根据特征成分图排序还原数据,根据 component 6 和 component 10 进行倒序显示。 从运行结果可以看到,正如所料,component 6 向右侧,component 10 是向左侧,通过 NMF 提取的特征更容易被人理解。
1 def nmf_test1(n):
2 person=datasets.fetch_lfw_people(min_faces_per_person=20)
3 #建立NMF模型,使用 150 个主要成分
4 nmf=NMF(150,random_state=2)
5 #将数据X转换成降维后的数据
6 componmets=nmf.fit_transform(person.data)
7 #按照所提取特征成分 components[n]倒序排列
8 index=np.argsort(componmets[:,n])[::-1]
9 #提取特征成分后还原数据
10 fix, axes1 = plt.subplots(3, 5, figsize=(60, 40))
11 for ax,index1 in zip(axes1.ravel(),index):
12 a = person.data[index1, :]
13 ax.imshow(person.data[index1, :].reshape(62, 47), cmap='viridis')
14 ax.set_title('original ' + str(index1), fontsize=100)
15 plt.show()
16
17 if __name__ == '__main__':
18 nmf_test1(6)
19 nmf_test1(10)运行结果
component 6

component 10

三、ML 流形学习
PCA、NMF 虽然是比较常用的降维算法,不过对非线性的数据集处理效果不太好,为了弥补这一缺陷 sklearn 提供了另一套方案 —— 流形学习 ML (manifold learning)。流形学习是一种无监督评估器,它尝试将一个低维度流形嵌入到一个高维度空间来描述数据集。 下面介绍几个常用的 ML 流形学习模型:MDS 多维标度法、LLE 局部线性嵌入法、Isomap 保距映射法、t-SNE 分布邻域嵌入算法。
3.1 MDS 多维标度法
多维标度法 MDS (multidimensional scaling)是最常用的流形学习模型之一,它可以把三维数据通过计算距离矩阵,投影到二维空间,还原最优的二维嵌入结果。它的基本特征是根据每个数据点与数据聚焦其他点的的距离来进行计算的。
构造函数
1 class MDS(BaseEstimator):
2 @_deprecate_positional_args
3 def __init__(self, n_components=2, *, metric=True, n_init=4,
4 max_iter=300, verbose=0, eps=1e-3, n_jobs=None,
5 random_state=None, dissimilarity="euclidean"):
6 self.n_components = n_components
7 self.dissimilarity = dissimilarity
8 self.metric = metric
9 self.n_init = n_init
10 self.max_iter = max_iter
11 self.eps = eps
12 self.verbose = verbose
13 self.n_jobs = n_jobs
14 self.random_state = random_state参数说明
- n_components:int, 默认值为2。int 时则是直接指定 NMF 降维后的特征维度数目,此时 n_components是一个大于等于 1 的整数。当使用默认值,即不输入n_components,此时n_components=min(样本数,特征数)。
- dissimilarity:可选值:{'euclidean', 'precomputed'}, 默认值为 'euclidean' 。用于定义模型的非类似性,euclidean:根据点与点之间的欧几里得距离进行计算; precomputed: 根据距离矩阵方式进行试算。
- metric:bool 类型,默认值为True,是否使用距离量度 MDS,False 使用非距离度量 SMACOF
- n_init:int 类型,默认值为 4,初始化 SMACOF 算法方式的运行次数。把应用的最小结果作为运行结果输出。
- max_iter:int 类型,默认值为 300 ,指定了模型优化的最大迭代次数。
- eps:float 类型,默认值为 1-e3,收敛阙值。
- verbose: int类型,默认值为0,代表详细程度。
- n_jobs:CPU 并行数,默认为None,代表1。若设置为 -1 的时候,则用所有 CPU 的内核运行程序。
- random_state:int 类型,默认None,随机数种子,推荐设置一个任意整数,同一个随机值,模型可以复现。
应用实例
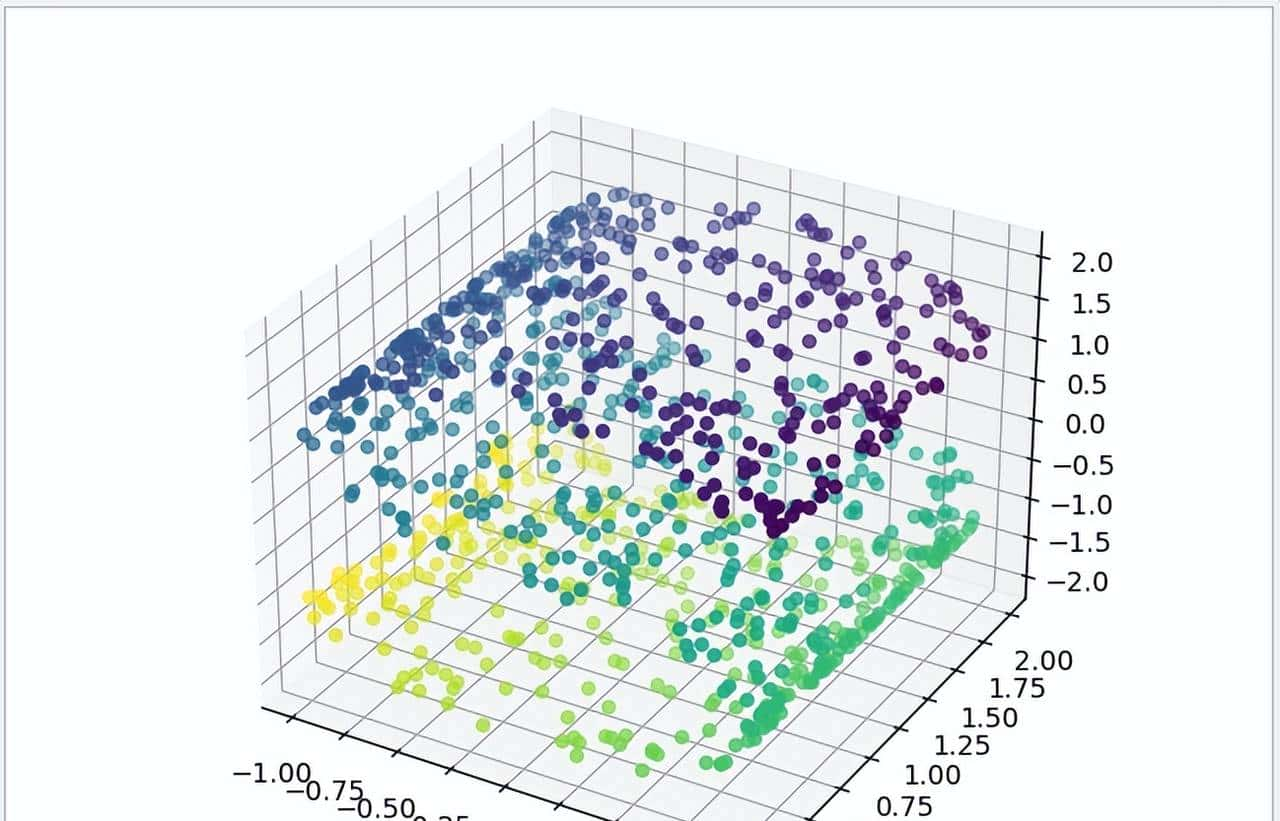
下面例子使用 sklearn 自带的测试数据集,此数据原来是三维空间中的立体数据,经过 MDS 二维评估转换后,投影会转换成可见的 S 形数据。
1 def mds_test():
2 # 测试数据
3 n_points = 1000
4 X, color = datasets._samples_generator.make_s_curve(n_points, random_state=0)
5 # 显示测试数据的3D图
6 ax1 = plt.axes(projection='3d')
7 ax1.scatter3D(X[:, 0], X[:, 1], X[:, 2], c=color)
8 plt.show()
9 # 初始化 MDS 模型
10 mds=MDS(n_components=2,random_state=1)
11 model=mds.fit_transform(X)
12 # 显示 MDS 评估结果
13 ax2=plt.axes()
14 plt.scatter(model[:,0],model[:,1])
15 plt.show()运行结果

3.2 LLE 局部线性嵌入法
使用 MDS 算法构建嵌入时,数据保留的是每对数据点之间的距离,不过在局部线性嵌入法模型 LLE (locally linear embedding) 的时候,数据不会保留所有的距离,而是保留邻节点间的距离,此值可通过参数n_neighbors 设定,默认为5 。
构造函数
1 class LocallyLinearEmbedding(TransformerMixin,
2 _UnstableArchMixin, BaseEstimator):
3 @_deprecate_positional_args
4 def __init__(self, *, n_neighbors=5, n_components=2, reg=1E-3,
5 eigen_solver='auto', tol=1E-6, max_iter=100,
6 method='standard', hessian_tol=1E-4, modified_tol=1E-12,
7 neighbors_algorithm='auto', random_state=None, n_jobs=None):
8 ......参数说明
- n_neighbors: int, 默认为 5 表明默认邻居的数量。
- n_components:int, 默认值为2。int 时则是直接指定降维后的特征维度数目,此时 n_components是一个大于等于 1 的整数。当使用默认值,即不输入n_components,此时n_components=min(样本数,特征数)。
- reg:float 类型,默认值 1e-3,正则化系数,当 n_neighbors大于n_components时,即近邻数大于降维的维数时,由于我们的样本权重矩阵不是满的,LLE通过正则化来解决这个问题。
- eigen_solver:str 类型, {'auto', 'arpack', 'dense'} 之一, 默认值 'auto' ,特征分解的方法。‘dense’:适合于非稀疏的矩阵分解; ‘arpack’: 虽然可以适应稀疏和非稀疏的矩阵分解,但在稀疏矩阵分解时会有更好算法速度。
- tol:float 类型,默认值为 1e-6,对当 eigen_solver 为 arpack 时的公差,代表求解方法精度。在 eigen_solver 为 dense 时无效。
- max_iter:int 类型,默认值为 100 ,指定了模型优化的最大迭代次数。
- method:str 类型 {'standard', 'hessian', 'modified', 'ltsa'} 之一, 默认值为 'standard' ,指定 LLE 的具体算法。一般来说 'hessian', 'modified', 'ltsa' 算法在同样的近邻数 n_neighbors 情况下,运行时间会比standard 的 LLE 长,当然降维的效果会稍微好一些。如果你对降维后的数据局部效果很在意,那么可以思考使用 'hessian', 'modified', 'ltsa' 或者增大n_neighbors,否则标准的 standard 就可以了。需要注意的是使用 modified 要求n_neighbors > n_components,而使用 hessian 要求n_neighbors > n_components * (n_components + 3) / 2
- hessian_tol:float 类型,默认值为 1e-4,当 method 为 hessian 时的公差精度,其他 method 无效
- modified_tol: float 类型,默认值为 1e-12,当 method 为 modified 时的公差精度,其他 method 无效
- neighbors_algorithm:str 类型,{'auto', 'brute', 'kd_tree', 'ball_tree'} 之一, 默认值为 'auto' ,代表 k近邻的实现方法,和KNN算法的使用的搜索方法类似。‘brute’ 对应第一种蛮力实现,‘kd_tree’对应第二种KD树实现,‘ball_tree’对应第三种的球树实现, ‘auto’则会在上面三种算法中做权衡,选择一个拟合最好的最优算法。需要注意的是,如果输入样本特征是稀疏的时候,无论我们选择哪种算法,最后scikit-learn都会去用蛮力实现 ‘brute’。
- n_jobs:CPU 并行数,默认为None,代表1。若设置为 -1 的时候,则用所有 CPU 的内核运行程序。
- random_state:int 类型,默认None,随机数种子,推荐设置一个任意整数,同一个随机值,模型可以复现。
使用与 MDS 一样的例子作为测试,把原来三维空间中的立体数据,经过 LLE 二维评估转换后,投影会转换成可见的 S 形数据。
1 def lls_test():
2 # 测试数据
3 n_points = 1000
4 X, color = datasets._samples_generator.make_s_curve(n_points, random_state=0)
5 # 显示测试数据的3D图
6 ax1 = plt.axes(projection='3d')
7 ax1.scatter3D(X[:, 0], X[:, 1], X[:, 2], c=color)
8 plt.show()
9 # 初始化 LLE 模型
10 lle=LLE(n_neighbors=299,n_components=2,method='standard',random_state=1)
11 model=lle.fit_transform(X)
12 # 显示 LLE 评估结果
13 ax2=plt.axes()
14 ax2.scatter(model[:,0],model[:,1])
15 plt.show()运行结果

3.3 Isomap 保距映射法
Isomap 模型与 LLE 模型类似,数据不会保留所有的距离,而是保留邻节点间的距离,此值可通过参数n_neighbors 设定,默认为5 。 但 Isomap 在现实使用中对高维数据源的学习效果比较差,但有比较好的嵌入效果,一般用于数据集的预处理,可以提供分析数据的线索。
构造函数
1 class Isomap(TransformerMixin, BaseEstimator):
2 @_deprecate_positional_args
3 def __init__(self, *, n_neighbors=5, n_components=2, eigen_solver='auto',
4 tol=0, max_iter=None, path_method='auto',
5 neighbors_algorithm='auto', n_jobs=None, metric='minkowski',
6 p=2, metric_params=None):
7 ......参数说明
- n_neighbors: int, 默认为 5 表明默认邻居的数量。
- n_components:int, 默认值为2。int 时则是直接指定降维后的特征维度数目,此时 n_components是一个大于等于 1 的整数。当使用默认值,即不输入n_components,此时n_components=min(样本数,特征数)。
- eigen_solver:str 类型, {'auto', 'arpack', 'dense'} 之一, 默认值 'auto' ,特征分解的方法。‘dense’:适合于非稀疏的矩阵分解; ‘arpack’: 虽然可以适应稀疏和非稀疏的矩阵分解,但在稀疏矩阵分解时会有更好算法速度。
- tol:float 类型,默认值为 1e-6,对当 eigen_solver 为 arpack 时的公差,代表求解方法精度。在 eigen_solver 为 dense 时无效。
- max_iter:int 类型,默认值为 None,当 eigen_solver 为 arpack 时,指定了模型优化的最大迭代次数。在 eigen_solver 为 dense 时无效。
- path_method: str 类型,{'auto', 'FW', 'D'} 之一, 默认值为 'auto'。 ‘FW’:使用 Floyd_Warshall 算法 ; ‘D’: 使用 Dijkstra 算法
- neighbors_algorithm:str 类型,{'auto', 'brute', 'kd_tree', 'ball_tree'} 之一, 默认值为 'auto' ,代表 k近邻的实现方法,和KNN算法的使用的搜索方法类似。‘brute’ 对应第一种蛮力实现,‘kd_tree’对应第二种KD树实现,‘ball_tree’对应第三种的球树实现, ‘auto’则会在上面三种算法中做权衡,选择一个拟合最好的最优算法。需要注意的是,如果输入样本特征是稀疏的时候,无论我们选择哪种算法,最后scikit-learn都会去用蛮力实现 ‘brute’。
- n_jobs:CPU 并行数,默认为None,代表1。若设置为 -1 的时候,则用所有 CPU 的内核运行程序。
- metric: str 类型 或 callable, 默认值为 ‘minkowski’。
- p: int 类型,默认值为2 。
- metric_params: dict类型 , 默认值 为 None,使用 metric 时的参数
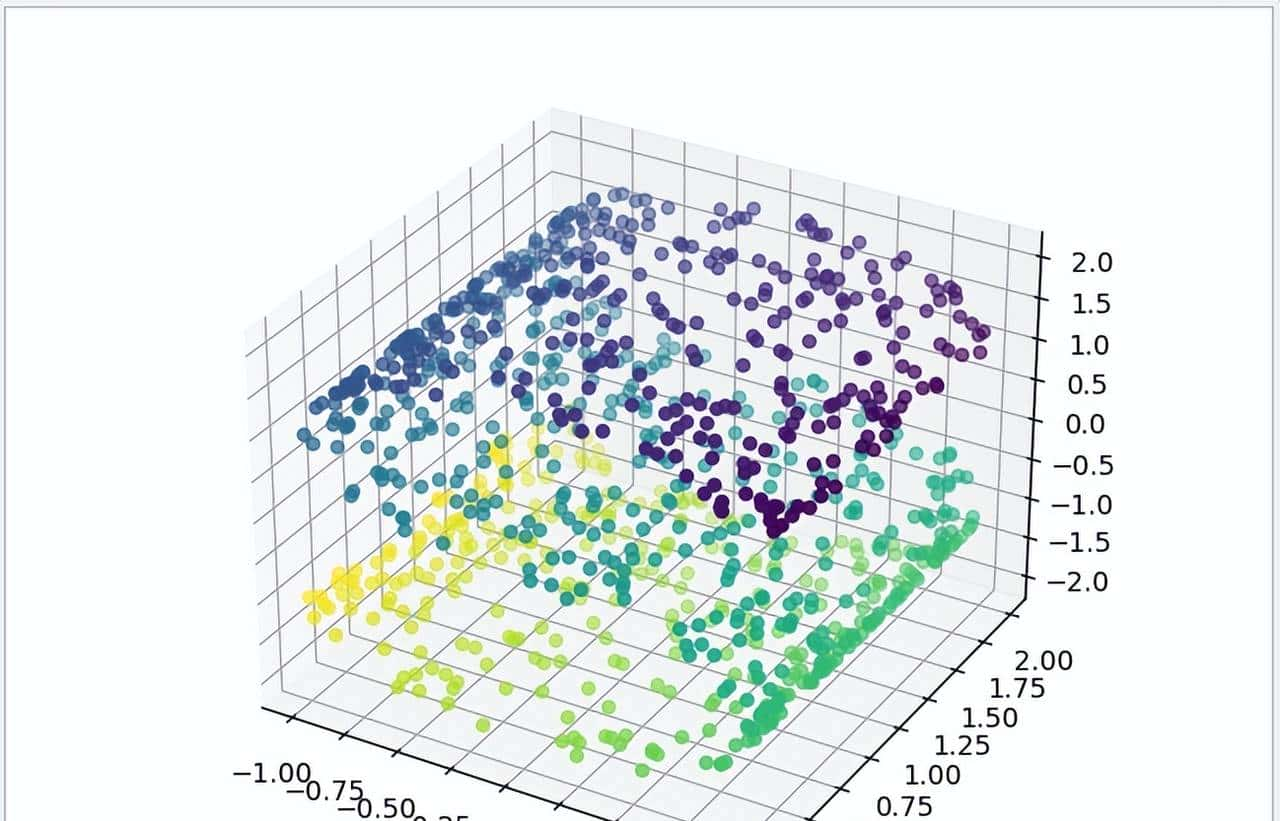
测试例子中使用 make_swiss_roll 数据集,使用 2000 个测试数据,把原来三维空间中的立体数据,经过 Isomap 二维评估转换后,观察其二维投影。
1 def iosmap_test():
2 # 测试数据
3 X, color =make_swiss_roll(n_samples=2000, noise=0.1)
4 # 显示测试数据的3D图
5 ax1 = plt.axes(projection='3d')
6 ax1.scatter3D(X[:, 0], X[:, 1], X[:, 2], c=color)
7 plt.show()
8 # 初始化 Isomap 模型
9 isomap =Isomap(n_neighbors=299,n_components=2)
10 model=isomap.fit_transform(X)
11 # 显示 Isomap 评估结果
12 ax2=plt.axes()
13 ax2.scatter(model[:,0],model[:,1])
14 plt.show()运行结果


3.4 t-SNE 分布邻域嵌入算法
t-SNE 分布邻域嵌入算法的思想是找到数据的一个二维表明方法,尽可能地分析数据点之间的距离,让原始特征中距离较近的点更加近,距离较远的点更加远。通过加大距离差值,更有利于分析特征差异。
构造函数
1 class TSNE(BaseEstimator):
2 @_deprecate_positional_args
3 def __init__(self, n_components=2, *, perplexity=30.0,
4 early_exaggeration=12.0, learning_rate=200.0, n_iter=1000,
5 n_iter_without_progress=300, min_grad_norm=1e-7,
6 metric="euclidean", init="random", verbose=0,
7 random_state=None, method='barnes_hut', angle=0.5,
8 n_jobs=None, square_distances='legacy'):参数说明
- n_components:int, 默认值为2。int 时则是直接指定降维后的特征维度数目,此时 n_components是一个大于等于 1 的整数。当使用默认值,即不输入n_components,此时n_components=min(样本数,特征数)。
- perplexity:float 类型,默认值为 30.0,较大的数据集一般需要更大的perplexity,一般介于5和50之间的值。
- early_exaggeration: float 类型,默认值为12.0 。控制原始空间中的自然簇在嵌入空间中的紧密程度以及它们之间的空间大小。对于较大的值,自然簇之间的空间将在嵌入空间中更大。同样,这个参数的选择不是很关键。如果在初始优化时成本函数增加,则早期夸大因子或学习率可能过高。
- learning_rate:float 类型,默认值为 200.0 表明学习率。学习率太高了,数据可能看起来像一个“球”形与它的最近邻大约相等的距离。如果是学习率太低了,大多数点可能看起来被压缩在一个密集的区域,几乎没有异常值,如果遇到此情形可尝试提高学习率可能会有所协助。
- n_iter:int 类型,默认值为1000,优化的最大迭代次数,一般应该在 200 以上。
- n_iter_without_progress:int 类型,默认值为 30,控制迭代停止条件。
- min_grad_norm:float 类型,默认值为 1e-7,如果梯度范数低于此阈值,则优化将被中止。
- metric:str 类型或 callable 默认值为 euclidean,表明计算特征数组中实例之间的距离时使用的度量为欧氏距离的平方。如果度量标准是字符串,则它必须是scipy.spatial.distance.pdist为其度量标准参数所允许的选项之一,或者是成对列出的度量标准.PAIRWISE_DISTANCE_FUNCTIONS。如果度量是“预先计算的”,则X被假定为距离矩阵。或者,如果度量标准是可调用函数,则会在每对实例(行)上调用它,并记录结果值。可调用应该从X中获取两个数组作为输入,并返回一个表明它们之间距离的值。
- init:str 类型,{'random', 'pca'}之一,或 narray 类型数组 (n_samples,n_components),默认值为 random,嵌入的初始化。PCA 初始化不能与预先计算的距离一起使用,一般比随机初始化更全局稳定。
- verbose:int 类型,默认值为 0,详细度等级。
- random_state: :int 类型,默认None,随机数种子,推荐设置一个任意整数,同一个随机值,模型可以复现。
- method:str 类型,默认值为 “barnes_hut”,默认情况下,梯度计算算法使用在O(NlogN)时间内运行的 Barnes-Hut 近似值。 method ='exact' 将运行在O(N ^ 2)时间内较慢但准确的算法上。当最近邻的误差需要好于3%时,应该使用准确的算法。但是,确切的方法无法扩展到数百万个示例。
- angle:float 类型,默认值为 0.5 。仅当 method ='barnes_hut' 时才有效,这是Barnes-Hut T-SNE的速度和准确性之间的折衷。 'angle'是从一个点测量的远端节点的角度大小(在[3]中称为theta)。如果此大小低于'角度',则将其用作其中包含的所有点的汇总节点。该方法对0.2-0.8范围内该参数的变化不太敏感。小于0.2的角度会迅速增加计算时间和角度,因此0.8会快速增加误差。
- n_jobs:int 类型,CPU 并行数,默认为None,代表1。若设置为 -1 的时候,则用所有 CPU 的内核运行程序。
- square_distances:True 或 'legacy‘,是否将距离值平方。’legacy‘ 意味着距离值只有在米制=“欧几里德” 时才平方。True表明所有度量的距离值都是平方的。
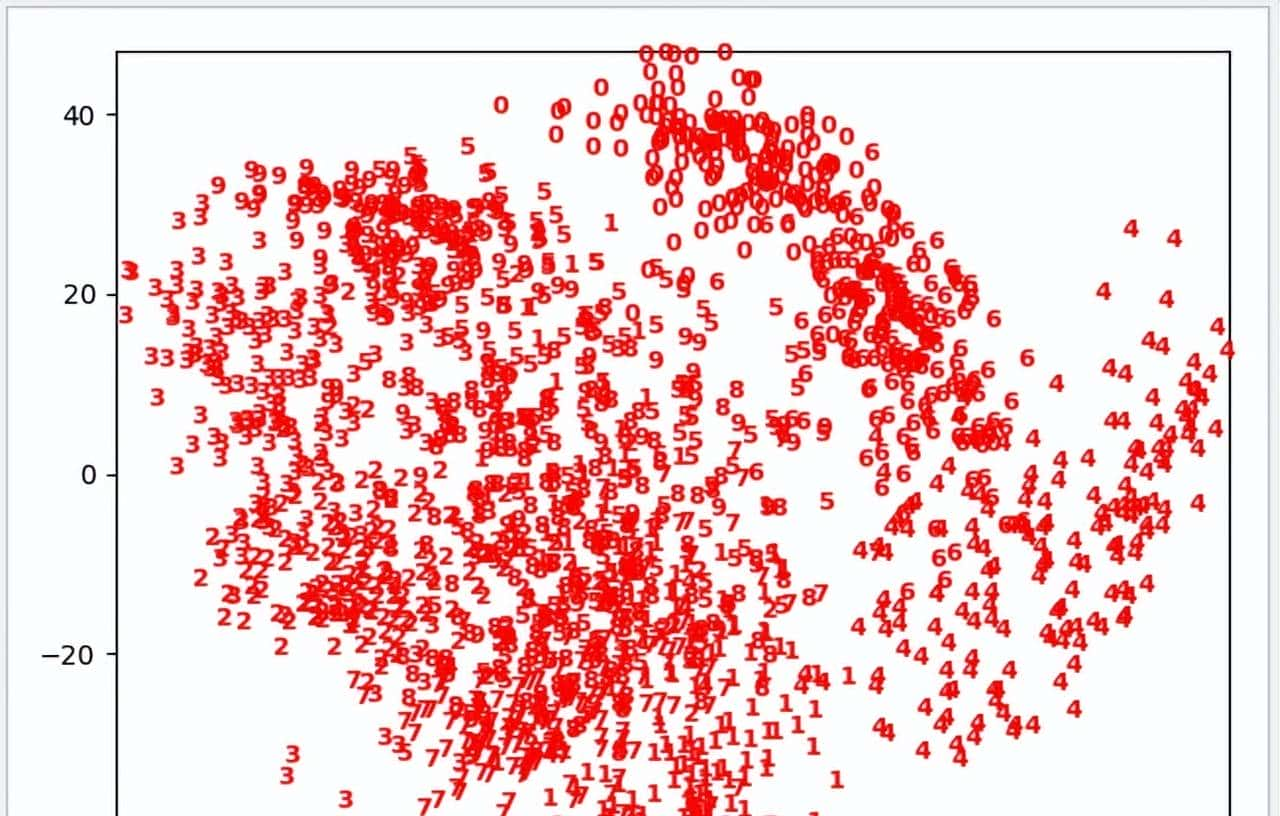
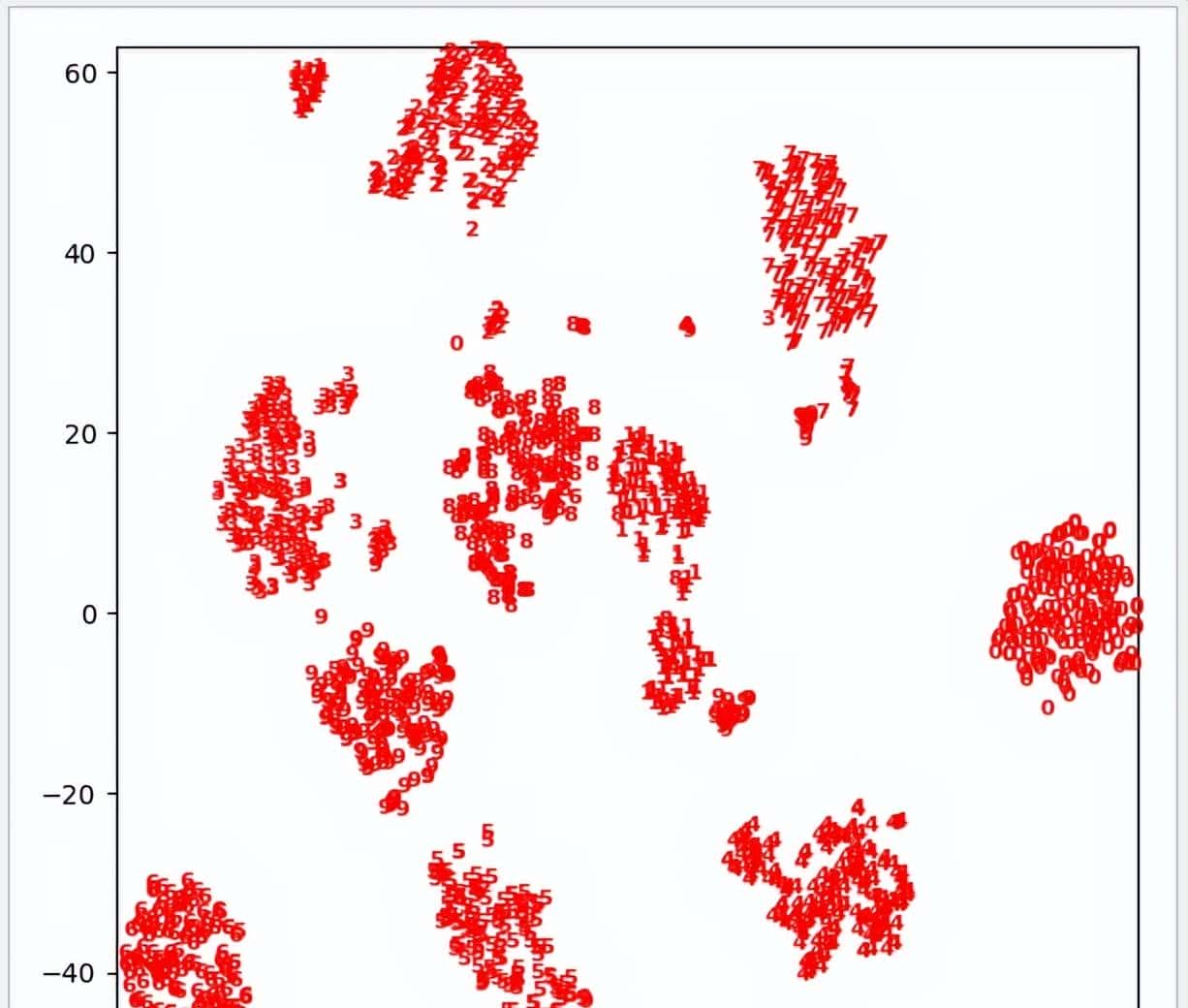
下面尝试使用 t-SNE 分析手写数字的数据集,由于 t-SNE 不支持变换新数据,所以它不包含 transform 方法,一般会直接使用 fit_transform 方法取代。用 digits 数据集来测试,使用 Isomap 模型测试时,尽管 0,4,6的数据比较有效地分开 ,然后其他大部分数据依然重叠得比较利害,测试结果末达到预期效果。使用 t-SNE 模型进行测试时,由于模型自身的特点,它可以把测试集的特征区别进一步放大,所以在测试结果中可以把 0~9 数字有效地区别开来。
1 def tsne_test():
2 # 输入测试数据
3 digits=load_digits()
4 # Isomap 模型测试
5 isomap=Isomap(n_neighbors=299,n_components=2)
6 model0=isomap.fit_transform(digits.data)
7 # 转出 Isomap 模型测试结果
8 plt.xlim(model0[:,0].min(),model0[:,0].max()+1)
9 plt.ylim(model0[:,1].min(),model0[:,1].max()+1)
10 for i in range(len(model0)):
11 plt.text(model0[i,0],model0[i,1],str(digits.target[i]),
12 color='red',fontdict={'weight':'bold','size':9})
13 plt.show()
14 # t-SNE 模型测试
15 tsne=TSNE(random_state=2,learning_rate=800)
16 model1=tsne.fit_transform(digits.data)
17 plt.figure(figsize=(6,6))
18 #输出 t-SNE 测试结果
19 plt.xlim(model1[:,0].min(),model1[:,0].max()+1)
20 plt.ylim(model1[:,1].min(),model1[:,1].max()+1)
21 for i in range(len(model1)):
22 plt.text(model1[i,0],model1[i,1],str(digits.target[i]),
23 color='red',fontdict={'weight':'bold','size':8})
24 plt.show()运行结果
Isomap 测试结果
t-SNE 测试结果
到此为此,本节介绍了4个常用的 ML 流形学习模型:MDS 多维标度法、LLE 局部线性嵌入法、Isomap 保距映射法、t-SNE 分布邻域嵌入算法。一般这些流形算法都用于在初期探索分析数据,但如果最终目标是监督学习的话则很少用到。
本章总结
由于篇幅关系,无监督学习中的数据集变换使用就先介绍到这里,关于聚类模型中 KMeans、DBSCAN、GMM 、Agglomerative 等模型将在 《 [Python 机器学习实战 —— 无监督学习(下)]》中详细讲述,敬请留意。
本章主要介绍了最常见的 PCA 主成分分析、NMF 非负矩阵分解等无监督模型,对 MDS 多维标度法、LLE 局部线性嵌入法、Isomap 保距映射法、t-SNE 分布邻域嵌入算法等 ML 流形学习模型的基础使用方法进行讲解,希望本篇文章对相关的开发人员有所协助。
在上篇《 [Python 机器学习实战 —— 无监督学习(上)]》介绍了数据集变换中最常见的 PCA 主成分分析、NMF 非负矩阵分解等无监督模型,举例说明使用使用非监督模型对多维度特征数据集进行降维的意义及实用方法。对 MDS 多维标度法、LLE 局部线性嵌入法、Isomap 保距映射法、t-SNE 分布邻域嵌入算法等 ML 流形学习模型的基础使用方法进行讲解。 本文将对聚类算法进行讲解,聚类算法就是将数据集划分成组的任务,这些组叫成簇,同一个簇内的数据点特征超级类似,不同簇内的数据点特征区别很大,这点与监督学习中的分类算法很类似,运行完成后系统会为同一簇内的数据分配同一个数字,不同簇的数字都不一样。常见的聚类模型有 KMeans、DBSCAN、GMM 、Agglomerative 等,下面将一一介绍。
四、KMeans 均值聚类
4.1 KMeans 的基本原理
KMeans 均值聚类是最简单最常用的聚类算法之一,它会尝试找到代表数据区域的簇中心,并保存在 cluster_centers 属性中。再把每个数据点分配给最接近的簇中心,并把每个簇中心设置为所分配数据点的平均值。当簇中心的值根据模型运算设置不再发生变化时,算法结束。
构造函数
1 class KMeans(TransformerMixin, ClusterMixin, BaseEstimator):
2 @_deprecate_positional_args
3 def __init__(self, n_clusters=8, *, init='k-means++', n_init=10,
4 max_iter=300, tol=1e-4, precompute_distances='deprecated',
5 verbose=0, random_state=None, copy_x=True,
6 n_jobs='deprecated', algorithm='auto'):- n_clusters:int 类型,默认为8,代表生成簇中心数目
- init:选择 {’k-means++’, ‘random ’} 之一, 或者传递一个ndarray向量,代表初始化方式,默认值为 ‘k-means++’。‘k-means++’ 用一种特殊的方法选定初始聚类中发,可加速迭代过程的收敛;‘random’ 随机从训练数据中选取初始质心。如果传递的是一个ndarray,则应该形如 (n_clusters, n_features) 并给出初始质心。
- n_init:int 类型,默认值为10,用不同的聚类中心初始化值运行算法的次数,最终解是在 inertia 意义下选出的最优结果。
- max_iter: int 类型,默认值为 300 ,代表模型优化的最大迭代数。
- tol:float类型,默认值为 1e-4 ,代表求解方法精度
- precompute_distances: 可选值 {‘auto’,True,False }, 代表预计算距离,计算速度更快但占用更多内存。‘auto’:如果 样本数乘以聚类数大于 12million 的话则不预计算距离;True:总是预先计算距离;False:永远不预先计算距离。’ deprecated ‘ 旧版本使用,新版已丢弃。
- verbose: int 类型,默认为 0,详细程度。
- random_state:默认值为None 随机数种子,推荐设置一个任意整数,同一个随机值,模型可以复现
- copy_x: bool 类型,默认值True。当 precomputing distances 生效时,将数据中心化会得到更准确的结果。如果把此参数值设为True,则原始数据不会被改变。如果是False,则会直接在原始数据上做修改并在函数返回值时将其还原。但是在计算过程中由于有对数据均值的加减运算,数据返回后,原始数据和计算前可能会有细小差别。
- n_jobs:int 类型,默认为 None, CPU 并行数。内部原理是同时进行n_init指定次数的计算时,若值为 -1,则用所有的CPU进行运算。若值为1,则不进行并行运算。若值小于-1,则用到的CPU数为(n_cpus + 1 + n_jobs)。因此如果 n_jobs值为-2,则用到的CPU数为总CPU数减1。’ deprecated ‘ 旧版本使用,新版已丢弃。
- algorithm: str 类型,{ auto,full,elkan } 三选一。full就是一般意义上的K-Means算法; elkan是使用的elkan K-Means算法; auto则会根据数据值是否是稀疏的(稀疏一般指是有大量缺失值),来决定如何选择full 和 elkan。如果数据是稠密的,就选择elkan K-means,否则就使用普通的Kmeans算法。
常用参数
- cluster_centers_: 代表簇中心 ,训练后簇中心都会被保存在此属性中
- labels_:返回数据点所属的簇标志,与 predict 方法返回值一样
4.2 KMeans 的应用场景
下面先用简单的 make_blobs 数据集了解 KMeans 的基础使用方式,KMeans 模型在分别把簇中心设置为 3 和 5,观察一下数据的变化。注意测试数据聚焦默认为 3 类,不过 KMeans 是无监督学习模型,不会使用数据集给出的结果进行分类,而是按照 n_clusters 的设置进行分类。这是 KMeans 模型的优点也可以说是缺点,虽然没有受数据集类型的限制,不过在运行前必须先设置簇的数量。实事上在现实场景中,许多数据事先是无法确定簇数量的。
1 def kmean_test(n):
2 #生成数据集
3 X, y = datasets.make_blobs(n_samples=100, n_features=2, random_state=1)
4 #使用KMeans模型,3个簇中心
5 kmean=KMeans(n_clusters=n)
6 model=kmean.fit_predict(X)
7 #显示运算结果
8 plt.scatter(X[:,0],X[:,1],s=50,c=model,marker='^')
9 plt.xlabel('feature0')
10 plt.ylabel('feature1')
11 #显示簇中心
12 center=kmean.cluster_centers_
13 plt.scatter(center[:,0],center[:,1],s=200,color='red',marker='.')
14 #显示簇边界
15 radii=[cdist(X[model==i],[center]).max() for i,center in enumerate(center)]
16 for c,r in zip(center,radii):
17 axes.add_patch(plt.Circle(c,r,alpha=0.3,zorder=1))
18 plt.show()
19
20 if __name__=='__main__':
21 kmean_test(3)
22 kmean_test(5)3 簇中心运行结果

5 簇中心运行结果

下面例子尝试利用 digits 数据集,通过 KMeans 训练,再查看一下分辨手写数字的准确率,可见单凭 KMeans 模型已经可以分辨手写数字的准确率到达将近80% 。
1 def kmean_test():
2 # 输入测试数据
3 digits=datasets.load_digits()
4 #使用KMeans模型,10个簇中心
5 kmean=KMeans(10)
6 #训练数据
7 model=kmean.fit_predict(digits.data)
8 #计算匹配度
9 labels=np.zeros_like(model)
10 for i in range(10):
11 mask=(model==i)
12 labels[mask]=mode(digits.target[mask])[0]
13 #计算准确率
14 acc=accuracy_score(digits.target,labels)
15 print(acc)运行结果

还记得在上一篇文章《 [Python 机器学习实战 —— 无监督学习(上)]》介绍 ML 流形学习中曾经提过, t-SNE 模型是一个非线性嵌入算法,特别擅长保留簇中的数据点。在此尝试把 KMeans 与 t-SNE 相结合一起使用,有意想不到的效果。 运行后可以发现末经过调试的算法,准确率已经可以达到 94% 以上。可见,只要适当地运用无监督学习,也能够精准地实现数据的分类。
1 def kmean_test():
2 # 输入测试数据
3 digits=datasets.load_digits()
4 #使用t-SNE 模型进行训练
5 tsne=TSNE()
6 model0=tsne.fit_transform(digits.data)
7 #使用KMeans模型进行训练
8 kmean=KMeans(10)
9 model1=kmean.fit_predict(model0)
10 #计算匹配度
11 labels=np.zeros_like(model1)
12 for i in range(10):
13 mask=(model1==i)
14 labels[mask]=model(digits.target[mask])[0]
15 #计算准确率
16 acc=accuracy_score(digits.target,labels)
17 print(acc)运行结果

4.3 通过核转换解决 KMeans 的非线性数据问题
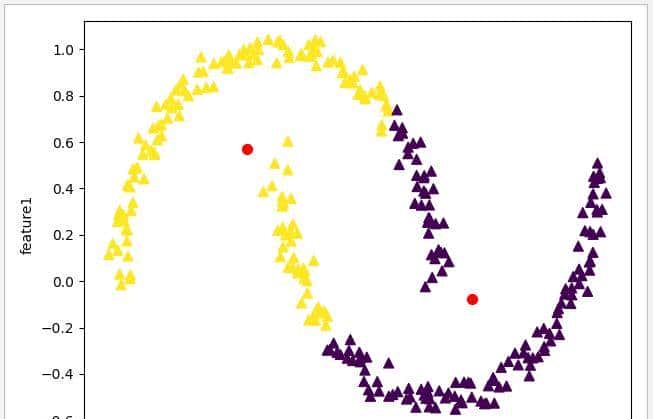
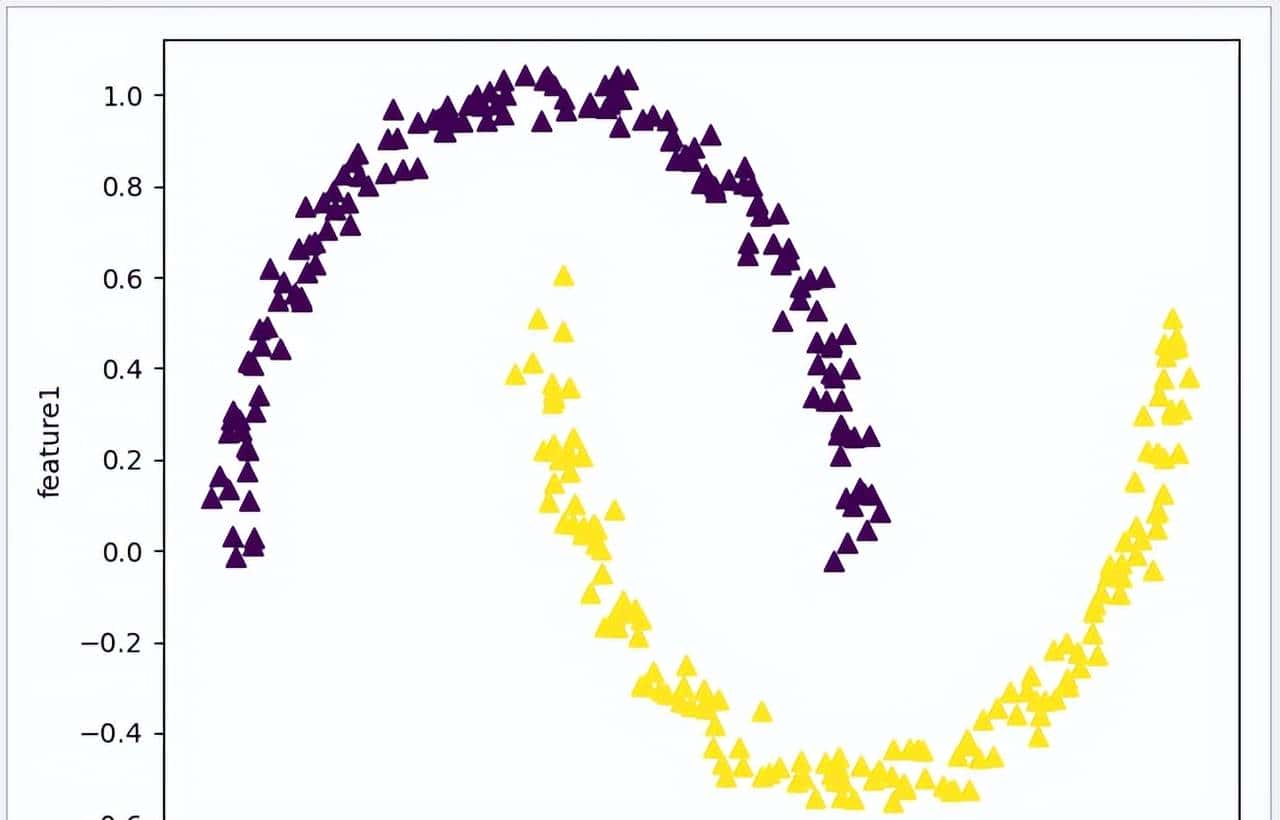
虽然 KMeans 模型适用的场景许多,不过 KMeans 也有一个问题就是它只能确定线性聚类边界,当簇中心点呈现非线性的复杂形状时,此算法是不起作用的。例如以 make_moons 数据集为例,把 noise 参数设置为 0.04,用 KMeans 模型进行测试。分别把 n_clusters 设置为 2 和 10,运行一下程序,可见单凭 KMeans 模型是无法分开复杂形状的非线性数据的。
1 def kmean_test(n):
2 #生成数据集
3 X, y = datasets.make_moons(n_samples=300, noise=0.04,random_state=1)
4 #使用KMeans模型,n个簇中心
5 kmean=KMeans(n_clusters=n)
6 model=kmean.fit_predict(X)
7 #显示运算结果
8 plt.scatter(X[:,0],X[:,1],s=50,c=model,marker='^')
9 plt.xlabel('feature0')
10 plt.ylabel('feature1')
11 #显示簇中心
12 center=kmean.cluster_centers_
13 plt.scatter(center[:,0],center[:,1],s=200,color='red',marker='.')
14 plt.show()
15
16 if __name__=='__main__':
17 kmean_test(2)
18 kmean_test(10)n_clusters 为 2 时
n_clusters 为 10 时

此时,大家可能会想起在上一章《 [Python 机器学习实战 —— 监督学习(下)]》中提到的核函数技巧,类似地 skLearn 也提供了 SpectralClustering 评估器来完成 KMeans 非线性边界的问题。
构造函数
1 class SpectralClustering(ClusterMixin, BaseEstimator):
2 @_deprecate_positional_args
3 def __init__(self, n_clusters=8, *, eigen_solver=None, n_components=None,
4 random_state=None, n_init=10, gamma=1., affinity='rbf',
5 n_neighbors=10, eigen_tol=0.0, assign_labels='kmeans',
6 degree=3, coef0=1, kernel_params=None, n_jobs=None,
7 verbose=False):- n_clusters:int 类型,默认为 8,代表生成簇中心数目
- eigen_solver: {'arpack', 'lobpcg', 'amg','None'} 之一,默认为 None ,代表特征值求解的策略 。
- n_components:int, 默认值为 None。int 时则是直接指定特征维度数目,此时 n_components是一个大于等于 1 的整数。当使用默认值,即不输入n_components,此时n_components=min(样本数,特征数)。
- random_state:int 类型,默认值为None, 随机数种子,推荐设置一个任意整数,同一个随机值,模型可以复现。
- n_init: int 类型,默认为值 10,当 assign_labels 为 ’kmeans' 时,指定聚类中心初始化值运行算法的次数。
- gamma:float 类型,默认值为1.0,指高斯核函数的中心值。如果用k近邻法,则此参数无用。
- affinity:str 类型 {’rbf','nearest_neighbors','precomputed' , 'precomputed_nearest_neighbors'} 之一 或 callable,默认值为 ‘rbf' ,用于设定构造矩阵的方法。 ‘ rbf ' 指使用径向基函数构造矩阵(RBF)高斯内核 ; 'nearest_neighbors' 指使用 k近邻算法来构造内核; 'precomputed' 是将X解释为一个预先计算的亲和力矩阵,较大值表明实例之间的类似性更大; 'precomputed_nearest_neighbors': 将X解释为稀疏图,从每个实例的n_neighbors的最近邻居构造一个二进制亲和矩阵。
- n_neighbors:int 类型,默认值为10。当 affinity 设定为 ‘nearest_neighbors’ 使用 k 近邻内核时的邻居数量,当使用 ‘rbf’ 高斯内核时无效。
- eigen_tol:float 类型,默认值为 0.0。 当`eigen_solver='arpack' 时候,设定拉普拉斯矩阵特征分解的停止判断依据。
- assign_labels:str 类型 {‘kmeans’, ‘discretize’} 之一,默认值为 ‘kmeans',用于指定分配标签的策略 。’kmeans‘ 是较常用的策略,初始化时灵敏性强。 ‘discretize’ 是随机初始化的另一种策略,灵敏性较弱。
- degree: int 类型,默认值为 3,当 eigen_solver 为 'arpack' 时,设定拉普拉斯矩阵特征分解的停止判据依据。
- coef0: float参数 默认为1.0,核函数中的独立项,控制模型受高阶多项式的影响程度, 只有对内核为 ‘poly’和‘sigmod’ 的核函数有用
- kernel_params: dict 类型 或者 str 类型,默认值为 None ,内核的参数。
- n_job:int 类型,默认值为 None 。当 affinity 为 ‘nearest_neighbors’ 和 ‘precomputed_nearest_neighbors’ 指定 CPU 并行数, 为 -1 指运行所有 CPU, 为 None 时只运行1个CPU。
- verbose: int 类型,默认为 0,详细程度。
尝试 SpectralClustering 评估器来完成非线性数据的分类问题,把 affinity 设置 'nearest_neighbors‘ 使用 k 近邻算法内核,assign_labels 设置为 'kmeans' 策略。从运行结果可以看到,SpectralClustering 使用 k 近邻内核完美地实现了数据边界的分离。
1 def kmean_test():
2 #生成数据集
3 X, y = datasets.make_moons(n_samples=300, noise=0.04,random_state=1)
4 #使用SpectralClustering评估器
5 spectral=SpectralClustering(n_clusters=2,affinity='nearest_neighbors',assign_labels='kmeans')
6 model=spectral.fit_predict(X)
7 #显示运算结果
8 plt.scatter(X[:,0],X[:,1],s=50,c=model,marker='^')
9 plt.xlabel('feature0')
10 plt.ylabel('feature1')
11 plt.show()运行结果
五、GMM 高斯混合模型
5.1 GMM 的基本原理
由于 KMeans 模型是围绕着簇中心而计算的,造成当中存在短板,数据点分配是依赖簇中心到数据点的平均值的,因此簇的模型必须是圆形的(在上节的例子中可以看出)。当簇中心比较接近时,数据点分配就会发生重叠而引起混乱。
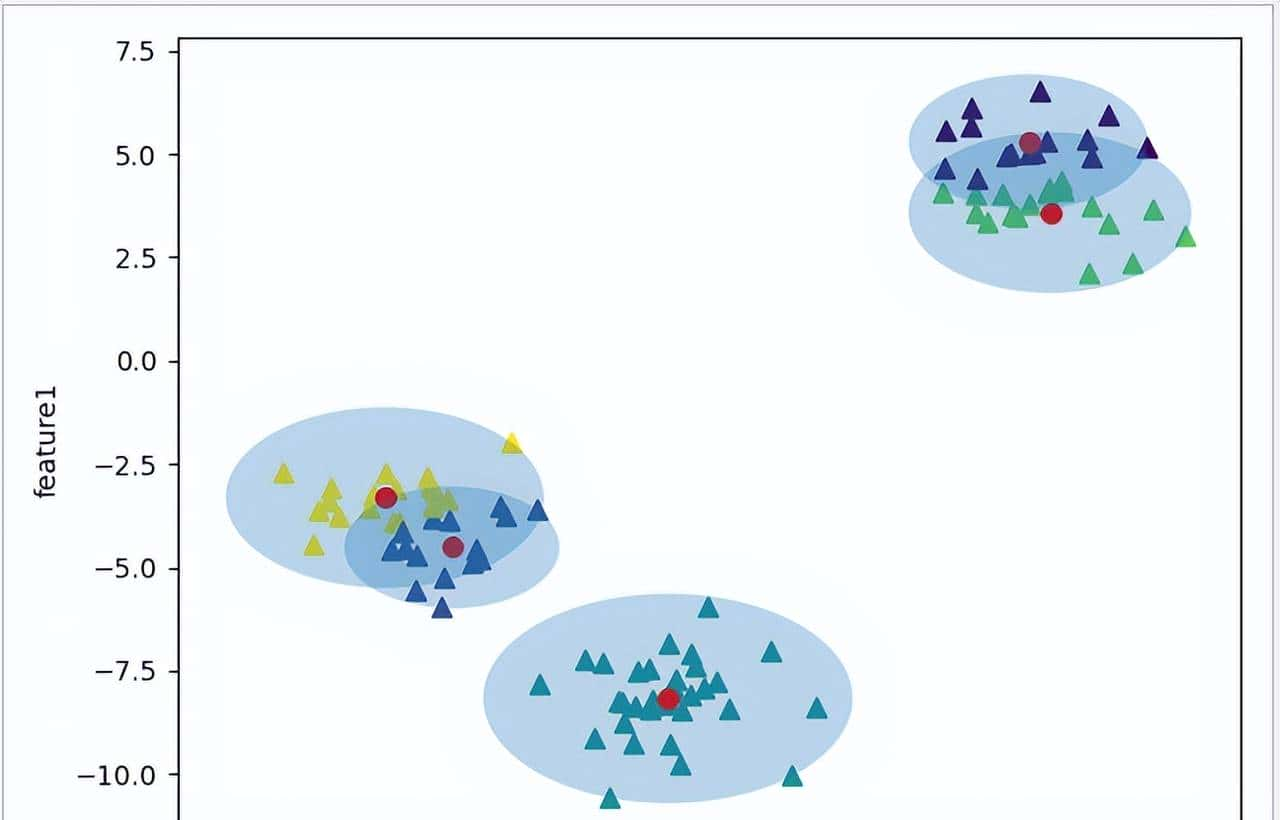
为解决 KMeans 模型的问题,GMM 高斯混合模型应运而生,它会改 KMeans 模型簇边界的计算方式,把圆形改成椭圆形,让数据边界更明显。
构造函数
1 class GaussianMixture(BaseMixture):
2 @_deprecate_positional_args
3 def __init__(self, n_components=1, *, covariance_type='full', tol=1e-3,
4 reg_covar=1e-6, max_iter=100, n_init=1, init_params='kmeans',
5 weights_init=None, means_init=None, precisions_init=None,
6 random_state=None, warm_start=False,
7 verbose=0, verbose_interval=10):- n_components: int 类型,默认为1,设定混合高斯模型个数
- covariance_type: str 类型 {‘full’,‘tied’, ‘diag’, ‘spherical ’} 之一,选择四种协方差类型,默认值为 ‘full’ 完全协方差矩阵。full: 对应完全协方差矩阵(元素都不为零), 每个分量都有各自的一般协方差矩阵 ; tied: 一样的完全协方差矩阵(HMM会用到)所有的分量都共享一样的一般协方差矩阵; ’ diag': 对角协方差矩阵(非对角为零,对角不为零)每个分量都有各自的对角协方差矩阵 ; ‘spherical': 球面协方差矩阵(非对角为零,对角完全一样,球面特性),每个组件都有它自己的单一方差。
- tol:float 类型,默认为1e-3,EM迭代停止阈值。
- reg_covar: float 类型,默认为 1e-6,协方差对角非负正则化,保证协方差矩阵均为正。
- max_iter: int 类型,默认值 100,最大迭代次数。
- n_init: int 类型,默认为1,初始化次数,指定聚类中心初始化值运行算法的次数。
- init_params: str 类型,{‘kmeans’, ‘random’} 之一,默认值 kmeans ,初始化参数实现方式
- weights_init: array 类型 (n_components, ) ,默认为 None ,用户提供的初始权重,若为空时,则使用 init _params 方法进行初始化。
- means_init: array 类型 (n_components, n_features), 默认为 None ,用户提供的初始权重,若为空时,则使用 init _params 方法进行初始化。
- precisions_init: array 类型 ,默认为 None ,用户提供的初始权重,若为空时,则使用 init _params 方法进行初始化。当 covariance_type 为 full 则为 (n_components, n_features, n_features) 格式; 'diag' 时格式为 (n_components, n_features) ; 'tied' 时格式为 (n_features, n_features); 'spherical' 时格式为 (n_components, )
- random_state :int 类型,默认值为None, 随机数种子,推荐设置一个任意整数,同一个随机值,模型可以复现。
- warm_start : bool 类型,默认为 False,若为True,则fit()调用会以上一次fit()的结果作为初始化参数,适合一样问题多次fit的情况,能加速收敛。
- verbose :int 类型,默认为0,使能迭代信息显示,可以为1或者大于1(显示的信息不同)
- verbose_interval :int 类型,默认10次,与 verbose 挂钩,若使能迭代信息显示,设置多少次迭代后显示信息。
参数说明
- weights_:array, 混合条件的权重
- means_:array, 均值
- covariances_: array 协方差阵
- converged_: bool 是否收敛
方法说明
- predict_proba(X):返回 [n_samples,n_clusters],预测给定的数据点属于某个簇的概率
- predict_proba() :预测所有数据点属于某个簇的概率
- score_samples(X):计算某个数据点样本的属于某个簇的加权对数概率
继续使用 make_blobs 数据集进行测试,适当修改数据点之间的间距,使用 GMM 模型进行计算,将为每个数据点找到对应每个簇的概率作为权重,然后更新每个簇的位置,将其标准化,最后把所有数据点的权重来确定形状。如此一来,数据点跟预期一样更为聚焦,数据边界会根据数据点的分布形成椭圆形。
1 def draw_ellipse(position, covariance, ax=None, **kwargs):
2 # 计算图形边界
3 if covariance.shape == (2, 2):
4 U, s, Vt = np.linalg.svd(covariance)
5 angle = np.degrees(np.arctan2(U[1, 0], U[0, 0]))
6 width, height = 2 * np.sqrt(s)
7 else:
8 angle = 0
9 width, height = 2 * np.sqrt(covariance)
10 # 画图
11 for nsig in range(1, 4):
12 ax.add_patch(Ellipse(position, nsig * width, nsig * height,
13 angle, **kwargs))
14
15 def gmm_test():
16 #测试数据集
17 X, y = datasets.make_blobs(n_samples=100,centers=4,n_features=2,random_state=1)
18 #修改数据点间距
19 rng=np.random.RandomState(12)
20 X1=np.dot(X,rng.randn(2,2))
21 fig,axes=plt.subplots(1,1)
22 #使用GMM模型
23 gmm=GaussianMixture(n_components=4,random_state=28)
24 model=gmm.fit_predict(X1)
25 #显示运算结果
26 plt.scatter(X1[:,0],X1[:,1],s=50,c=model,marker='^')
27 plt.xlabel('feature0')
28 plt.ylabel('feature1')
29 #显示簇边界
30 n=0.2/gmm.weights_.max()
31 for mean,covar,weight in zip(gmm.means_,gmm.covariances_,gmm.weights_):
32 draw_ellipse(mean,covar,ax=axes,alpha=weight*n)
33 plt.show()
34
35 if __name__=='__main__':
36 gmm_test()运行结果

六、Agglomerative 凝聚聚类
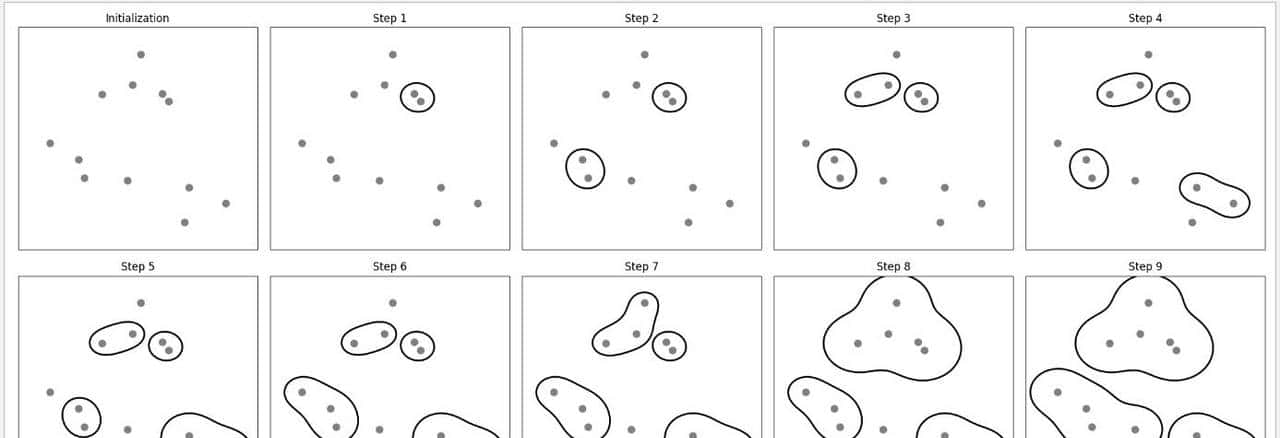
Agglomerative 凝聚聚类算法第一会声明每个点都是一个簇,然后合并两个最类似的簇,直到满足设定条件准则为止。这个准则可以通过 linkage 参数确定,linkage 为 { 'ward', 'complete', 'average', 'single' } 之一,下面将会详细说明。 观察下面系统自带的合并过程,当运行到第4步后,两点最相近的簇完成合并,到第5步一个两点簇增加到三个点,当三个点的簇完成合并后,到第8步,五点的簇开始合并,如此类推,到最后完成合并任务。
构造函数
1 class AgglomerativeClustering(ClusterMixin, BaseEstimator):
2 @_deprecate_positional_args
3 def __init__(self, n_clusters=2, *, affinity="euclidean",
4 memory=None,
5 connectivity=None, compute_full_tree='auto',
6 linkage='ward', distance_threshold=None,
7 compute_distances=False):- n_clusters:int 类型,默认值为2,指定最终分类簇的数量
- affinity:str 类型,{ ’euclidean’,’l1’,’l2’,’mantattan’,’cosine’,’precomputed’ }之一,默认值为 ’euclidean’ ,作用是选择一个计算距离的可调用对象。当 linkage=’ward’,affinity 只能为’euclidean’
- memory:str 类型,默认值为 None, 用于缓存计算结果的输出,默认不作任何缓存。如果设定字符串路径,则它会缓存到对应的路径。
- connectivity:array-like 或 callable 类型,默认为 None,用于指定连接矩阵,临近的数据将使用一样的结构。
- compute_full_tree:bool 类型,默认值为 auto,当训练了 n_clusters后,训练过程就会停止,但是如果 compute_full_tree=True,则会继续训练从而生成一颗完整的树。当 distance_threshold 不为 None 时,则 compute_full_tree 必须为是 True。
- linkage:str 类型, { 'ward', 'complete', 'average', 'single' } 之一,默认值为 'ward' 一个字符串,用于指定链接算法。‘ward’:单链接 single-linkage,使所有簇中的方差 dmindmin 增加最小的簇合并; ‘complete’:全链接 complete-linkage 算法,使簇中点之间的最大距离 dmaxdmax 最小的两个簇合并 ; 'average’:均连接 average-linkage 算法,使簇中所有点之间平均距离 davgdavg 最小的两个簇合并。’single' :新增于 0.20 版,使簇中所有点距离最小的两个簇合并。
- distance_threshold:float 类型,默认值为 None,链接距离阈值超过此设置,集群将不会合并。当使用此设置时,‘n_clusters’ 必须是 None 和 'compute_full_tree' 必须是 True 。
- compute_distances:bool 类型,默认值为 False,当不使用 distance_threshold 时,计算簇之间的距离。此功能可以用于使树状图可视化,但此计算将消耗计算机的内存。
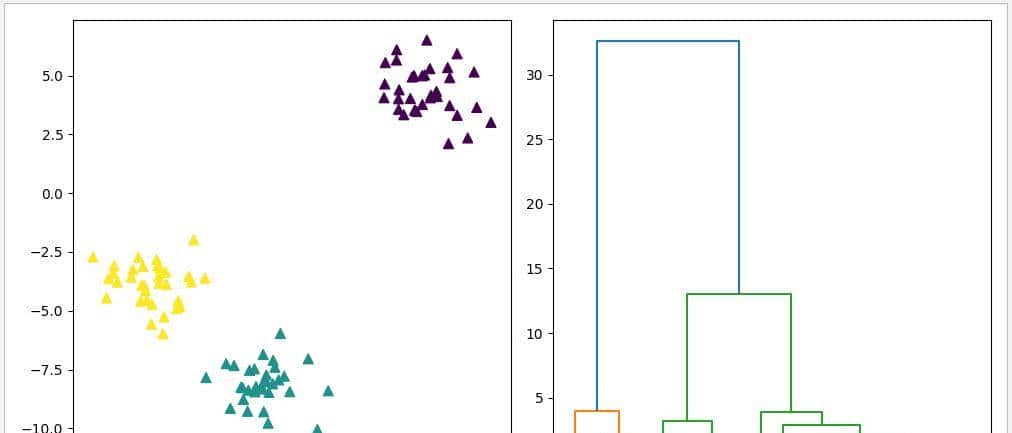
使用 make_blobs 数据集作测试,把 n_cluster3 设置为 3,由于 AgglomerativeClustering 没有 predict 方法,可以直接使用 fit_predict 方法进行计算。完成计算后,可以利用 SciPy 的 dendrogram 函数绘制树状图,以观察簇的构建过程。
1 def agglomerative_test():
2 # 测试数据集
3 X,y=make_blobs(random_state=1)
4 # 使用 AgglomerativeClustering模型
5 agglomerative=AgglomerativeClustering(n_clusters=3)
6 # 开始运算
7 model=agglomerative.fit_predict(X)
8 # 显示簇合并结果
9 fig, axes = plt.subplots(1, 2, figsize=(10,5))
10 ax0=axes[0]
11 ax0.scatter(X[:,0],X[:,1],s=50,c=model,marker='^')
12 # 显示树状图
13 ax1=axes[1]
14 shc.dendrogram(shc.ward(X[::5]))
15 plt.show()运行结果
七、DBSCAN 密度聚类
DBSCAN 密度聚类是最常用的模型之一,一般用于分析形状比较复杂的簇,还可找到不属于任何簇的数据点。上面介绍到的 KMeans 模型、SpectralClustering 评估器、Agglomerative 模型都需要在建立模型前通过 n_clusters 参数预先设置簇的个数,不过在复杂的数据中,单凭简单的数据分析去正确评估簇数据是很困难的。 DBSCAN 的优点在于它不需要预先设置簇的个数,而是通过特征空间的数据点密度来区分簇。DBSCAN 最常用到的参数是 eps 和 min_samples,eps 是用于设定距离,DBSCAN 将彼此距离小于 eps 设置值的核心样本放在同一个簇。min_samples 则用于设置同一簇内数据点的最少数量,如果数据点数量少于此设置则把这此点默认为噪点 noise,数据点数量大于等于此数值时则默认一个簇。
1 class DBSCAN(ClusterMixin, BaseEstimator):
2 @_deprecate_positional_args
3 def __init__(self, eps=0.5, *, min_samples=5, metric='euclidean',
4 metric_params=None, algorithm='auto', leaf_size=30, p=None,
5 n_jobs=None):- esp:float 类型,默认值为 0.5,用于设定距离,DBSCAN 将彼此距离小于 eps 设置值的核心样本放在同一个簇。
- min_samples: int 类型,默认值为5,用于设置同一簇内数据点的最少数量,如果数据点数量少于此设置则把这此点默认为噪点 noise,数据点数量大于等于此数值时则默认一个簇。
- metric:string 或 callable 类型,['cityblock', 'cosine', 'euclidean', 'l1', 'l2', 'manhattan'] 之一,默认值为 ‘euclidean’, 用于定义计算数据点之间的距离时使用的度量。使用欧式距离 “euclidean”; 使用曼哈顿距离 “manhattan”;
- metric_params: dict 类型,默认值为 None,根据 metric 选择填入相关参数。
- algorithm:str 类型,{'auto', 'ball_tree', 'kd_tree', 'brute'} 之一,默认值为 auto,定义计算近邻数据点的方法。 ‘auto’:会在上面三种算法中做权衡,选择一个拟合最好的最优算法。‘ball_tree’ 球面树搜索,数据点比较分散时可试用 ball_tree; kd_tree: kd树搜索,数据点分布比较均匀时效率较高; ‘brute’:暴力搜索;
- leaf_size:int 类型,默认为30,定义停止建立子树的叶子节点数量的阈值。 最近邻搜索算法参数,为使用KD树或者球树时, 这个值越小,则生成的KD树或者球树就越大,层数越深,建树时间越长,反之,则生成的KD树或者球树会小,层数较浅,建树时间较短。
- p: 最近邻距离度量参数。只用于闵可夫斯基距离和带权重闵可夫斯基距离中p值的选择,p=1为曼哈顿距离, p=2为欧式距离。如果使用默认的欧式距离不需要管这个参数。
- n_jobs:CPU 并行数,默认为None,代表1。若设置为 -1 的时候,则用所有 CPU 的内核运行程序。
使用 make_blobs 数据集,分别把 eps 设置为 0.5,1.0,1.5,观察一下测试结果,紫蓝色的是噪点。随着 eps 的增大,噪点越来越少,测试结果也越来越接近真实数据。不过 eps 对结果的影响也很大,如果 eps 设置得过大,可能会导致所有数据点形成同一个簇,如果 eps 太小,可能会导致所有数据点都是噪点。
1 def dbscan_test(eps,title):
2 # 测试数据集
3 X,y=datasets.make_blobs(random_state=1)
4 # 使用DBSCAN,输入eps参数
5 dbscan=DBSCAN(eps=eps)
6 model=dbscan.fit_predict(X)
7 # 显示测试后结果
8 plt.scatter(X[:,0],X[:,1],c=model,s=50,marker='^')
9 plt.xlabel('feature0')
10 plt.ylabel('feature1')
11 plt.title(title)
12 plt.show()
13
14 if __name__=='__main__':
15 dbscan_test(0.5,'EPS:0.5 ')
16 dbscan_test(1,'EPS 1')
17 dbscan_test(1.5,'EPS 1.5')运行结果
eps = 0.5

eps = 1.0

eps = 1.5

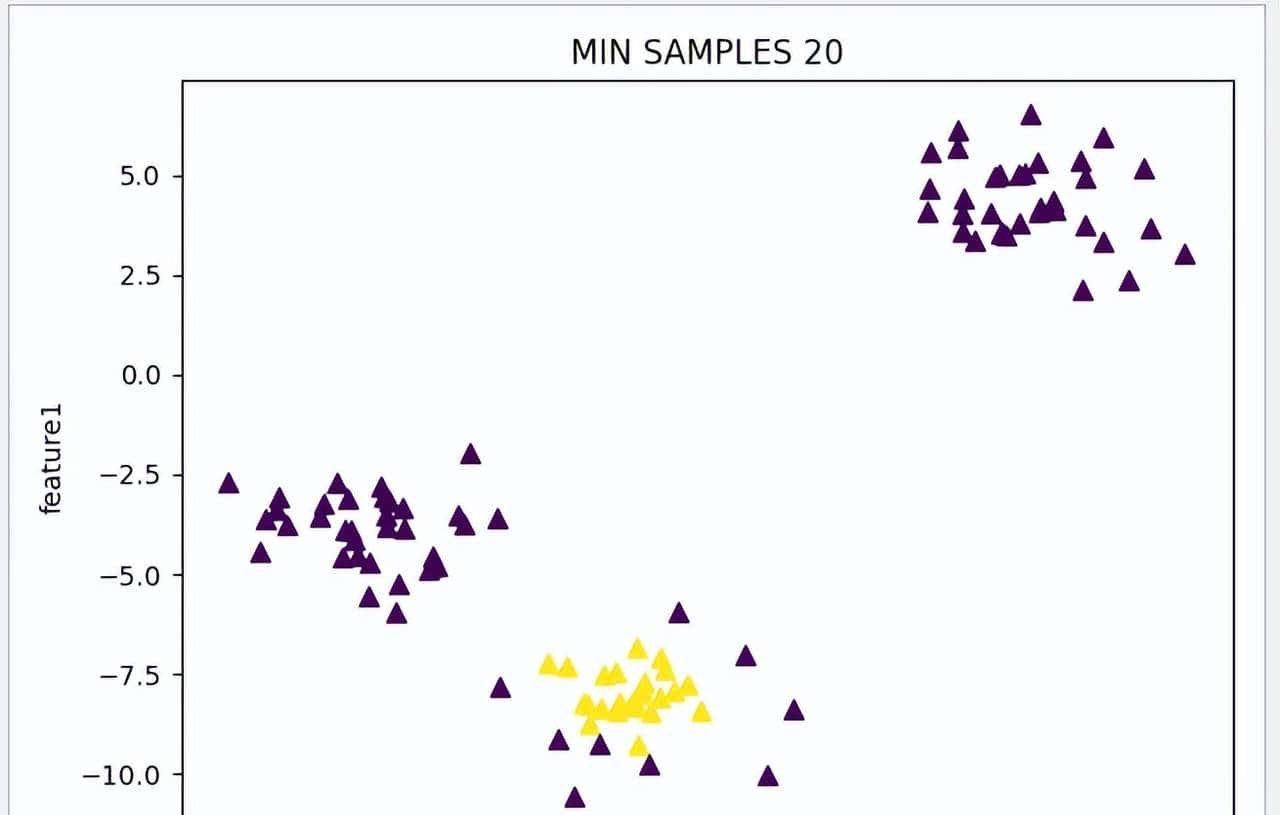
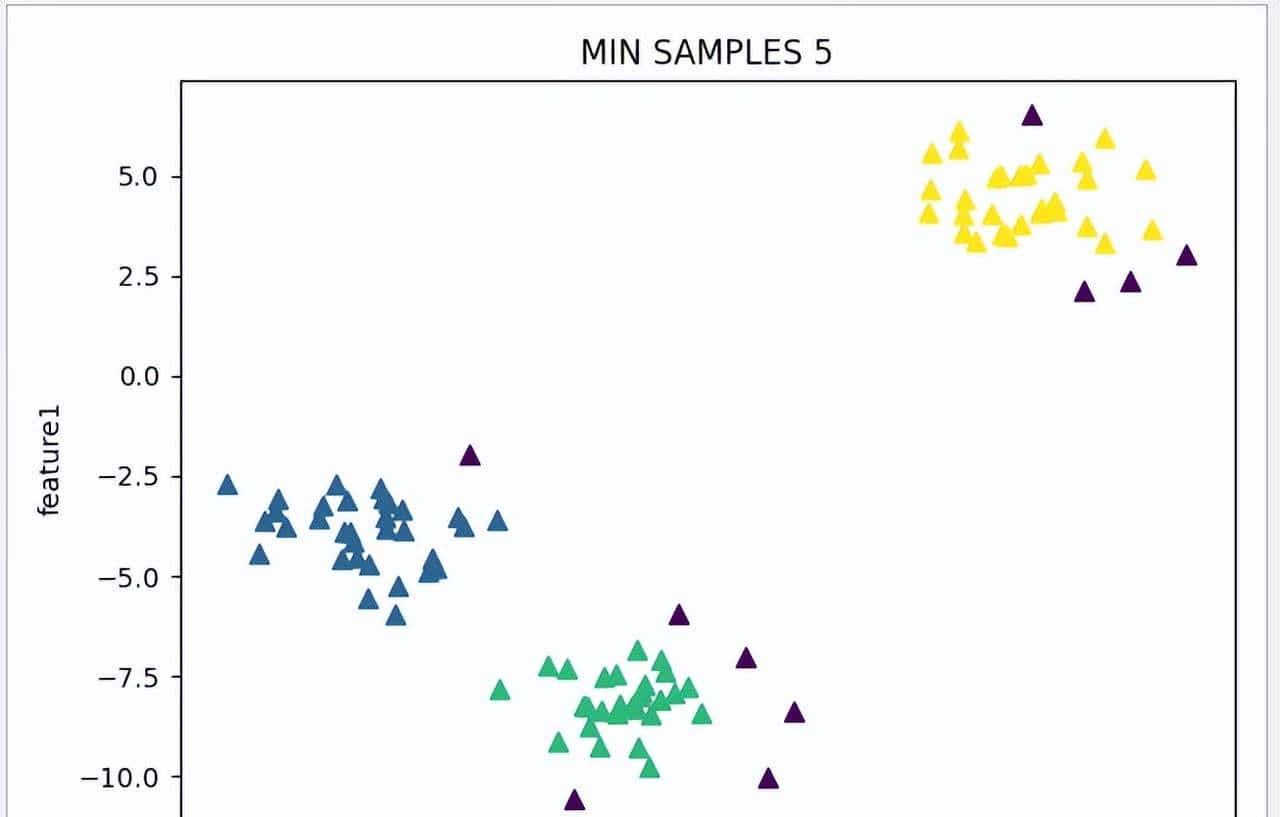
使用一样数据集,把 eps 设置为1.0,尝试修改 min_samples 参数,看看参数对模型的影响,紫蓝色为噪点。随着 min_samples 的减少,测试结果也越来越接近真实数据。
1 def dbscan_test(min,title):
2 # 测试数据集
3 X,y=datasets.make_blobs(random_state=1)
4 # 使用DBSCAN,输入eps,min_samples参数
5 dbscan=DBSCAN(eps=1.0,min_samples=min)
6 model=dbscan.fit_predict(X)
7 # 显示测试后结果
8 plt.scatter(X[:,0],X[:,1],c=model,s=50,marker='^')
9 plt.xlabel('feature0')
10 plt.ylabel('feature1')
11 plt.title(title)
12 plt.show()
13
14 if __name__=='__main__':
15 dbscan_test(20,'MIN SAMPLES 20')
16 dbscan_test(10,'MIN SAMPLES 10')
17 dbscan_test(5,'MIN SAMPLES 5')运行结果
min_samples = 20
min_samples = 10

min_samples = 5
第四节介绍 KMeans 模型时曾经介绍到 KMeans 模型是无法分开复杂形状的非线性数据的,只能用到 SpectralClustering 评估器来解决此类问题。不过 DBSCAN 模型的密度分布算法侧可以轻松地解决此类问题,同样使用 make_moons 数据集,使用 DBSCAN 模型时把 eps 设置为0.4,min_samples 设置为30。通过测试结果可以看出,只需要利用 DBSCAN 模型最常用的参数配置就可以解决复杂的非线性数据问题。
1 def dbscan_test(title):
2 # 测试数据集
3 X, y = datasets.make_moons(n_samples=300, noise=0.04,random_state=1)
4 # 使用DBSCAN,输入eps,min_samples参数
5 dbscan=DBSCAN(eps=0.4,min_samples=30)
6 model=dbscan.fit_predict(X)
7 # 显示测试后结果
8 plt.scatter(X[:,0],X[:,1],c=model,s=50,marker='^')
9 plt.xlabel('feature0')
10 plt.ylabel('feature1')
11 plt.title(title)
12 plt.show()
13
14 if __name__=='__main__':
15 dbscan_test('DBSCAN')运行结果

在前一章《 [Python 机器学习实战 —— 无监督学习(上)]》中介绍到的 PCA 模型, 下面这个例子就是先利用PCA模型对数据特征进行降维处理,然后再使用 DBSCAN 找出噪点,观察一下噪点与簇数据的区别出自哪里。使用 fetch_lfw_person 数据集,利用 PCA 模型把主要成分保持在 95%,完成训练后再使用 DBSCAN 模型,把 eps 设置为 23,min_samples 设置 3 ,再进行训练。 最后把噪点通过主要成分还原数据进行显示,从运行结果可以看到,噪点主要是由于人物有带帽子,张开嘴巴,用手捂嘴等动作造成的。 通过噪点分析,能找出许多有趣的与别不同的特征。
1 def dbscan_test():
2 # 测试数据集
3 person = datasets.fetch_lfw_people()
4 # 使用PCA模型把特征降至60
5 pca=PCA(0.95,whiten=True,random_state=1)
6 pca_model=pca.fit_transform(person.data)
7 # 使用DBSCAN,输入eps,min_samples参数
8 dbscan=DBSCAN(eps=23,min_samples=3)
9 model=dbscan.fit_predict(pca_model)
10 # 显示测试后结果
11 fig,axes=plt.subplots(5, 5, figsize=(25,25))
12 # 获取噪点的特征成分图
13 noise=pca_model[model==-1]
14 # 输出噪点数量
15 print(str.format('noise len is {0}'.format(len(noise))))
16 for componemt,ax in zip(noise,axes.ravel()):
17 # 获取噪点提取成分后还原图
18 image=pca.inverse_transform(componemt)
19 ax.imshow(image.reshape(62, 47), cmap = 'viridis')
20 plt.show() 运行结果


本章总结
本章主要介绍了 KMeans、GMM 、Agglomerative 、DBSCAN 等模型的使用,KMeans 是最常用最简单的模型,它尝试根据 n_clusters 设置找到代表数据区域的簇中心。而 GMM 可以看成是升级版的 KMeans ,它会改 KMeans 模型簇边界的计算方式,把圆形改成椭圆形,让数据边界更明显。Agglomerative 则更类似于树模型,使用近邻合并的模型,把相近的数据点合并为簇。DBSCAN 是更智能化的模型,通过数据点的聚集程度判断簇中心,在没有设置固定 n_clusters 的情况下分配出符合实际情况的簇。
对机器学习的原理及应用场景介绍到这里结束,后面将开始讲述深度学习的相关内容,敬请留意。



















- 最新
- 最热
只看作者